Peptides. Séquençage et détermination du pHi pour orienter la séparation par chromatographie et électrophorèse (Exercices)
Téléchargement de fichiers en Biochimie SUPPORTS

Les peptides résultant de l'hydrolyse des protéines couvrent les dipeptides (avec deux acides aminés) et des oligopeptides. Leur ionisation dépend de l'ionisation des acides aminés les composant. Ceci définit leur sens de migration en électrophorèse ou leur ordre d'élution en chromatographie d'échange d'ions. La détermination de séquences des peptides fait appel à l'hydrolyse acide, l'hydrolyse enzymatique (trypsine, chymotrypsine, aminopeptidase, ..)
Peptides particuliers. Aspartame et glutathion
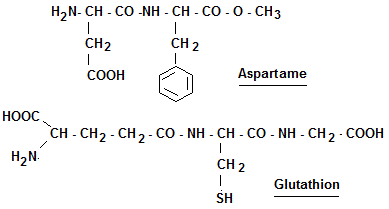
Pour chacune de ces molécules, indiquer les acides aminés les composant et les liaisons peptidiques et peptidoïdes (pseudopeptidiques). Quelles particularités présentent les deux molécules. L'aspartame a un pouvoir sucrant très supérieur à celui du saccharose. Il est utilisé comme édulcorant. Le glutathion est un antioxydant.
-
Aspartame. Il est constitué de l'acide aspartique, la phénylalanine
et le méthanol. C'est l'Aspartyl-Phénylalanine-méthylester.
-
Glutathion. Il est constitué des acides aminés Glu, Cys
et Gly. C'est le gamma-glutamyl-cystéyl-glycine. La liaison entre
Glu et Cys est une liaison peptidoïque particulière à laquelle participe le carboxyle de Glu situé sur le carbone gamma (carbone 4), au lieu du carbone 2 (carbone alpha). Par contre, la liaison entre Cys et Gly est une liaison peptidique (vraie).
Soit
le pentapeptide suivant: Lys-Val-His-Glu-Met.
- Ecrire la formule chimique développée de ce peptide.
- Etudier la variation de sa charge nette en fonction du pH et en déduire
la valeur de son pH isoélectrique.
Données: pK des différents groupements ionisables
- Formule chimique développée du pentapeptide
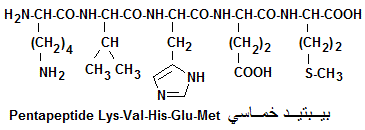
- Ordre de dissociation du pH le plus faible au pH le plus fort: COOH de Met (pK1) -> COOH radical de Glu (pK2) -> NH radical de His (pK3) -> NH2 de Lys (pK4)-> NH2 radical de Lys (pK5).
P+++ ..pK1..P+++- ..pK2..PP+++-- ..pK3..P++-- ..pK4..P+-- ..pK5..P--
Le pHi du pentapeptide est la moyenne des pK qui entoure la forme à charge nette égale à zéro, soit la moyenne de pk3 et pK4. Donc pHi = (6+8,95)/2 = 7,48
Ionisation d'un mélange de péptides et séparation par chromatographie et électrophorèse
Soit
un mélange de trois peptides:
- A: Glu-Cys-Leu-Glu,
- B: Arg-Pro-Gly-Lys,
- C: Val-Ile-Ile-Gly
1/ Le mélange est soumis à une chromatographie échangeuse
de cations. L'élution est faite par gradient de pH de 1 à
10. Indiquer l'ordre d'élution des trois peptides.
2/ Le même mélange est soumis à une électrophorèse à pH = 7,0. Représenter par un schéma l'électrophorégramme. Justifier.
-
1/ En chromatographie échangeuse de cations (+), la charge de la
résine est négative (-), les ions fixés sont des
cations. Les peptides intimement fixés sur le résine sont
ceux à pHi plus élevé. Le pHi du peptide A (3,22)
est inférieur à ceux des peptides C (5,98) et peptide B
(11,5). Lors de l'élution, l'augmentation du pH à plus de
3,22 (pHi de A) fait changer la charge électrique de A, devenue
négative. Il en résulte une répulsion avec la charge
négative de la résine. L'ordre d'élution des trois
peptides est A, C, B.
- 2/ Electrophorégramme: Les pHi des trois peptides encadre
le pH 7,0 avec des marges plus larges. De ce fait, le dépôt
du mélange sera fait au centre du support de migration. A pH =
7,0, le peptide A sera chargé négativement et migrera vers
l'anode (+). La charge du peptide C (pHi = 5,98) sera légèrement
négative et migrera peu. La charge électrique du peptide
B (pHi = 11,5) sera hautement positive et migrera vers la cathode (-)
Détermination de la séquence en acides aminés d'un tétrapeptide
Après
hydrolyse partielle par la trypsine d'un polypeptide, un tétrapeptide copmpsé des acides aminés Tyr, Arg, Cys, Glu, a été isolé.
L'acide aminé N-terminal a un rôle important dans la formation des ponts covalents interchaînes et intrachaînes des protéines.
L'acide aminé suivant migre le plus près de l'anode à pH = 8,5 par rapport aux autres acides aminés de ce peptide
Donner la séquence du tétrapeptide
Séquence du tétrapeptide: La formation de ponts covalents inter et intrachaînes est assurée par la Cystéine (Cys). Elle est donc le premier acide aminé côté N-terminal. L'acide aminé migrant plus près de l'anode (+) à pH 8,5 ne peut être que Glu (=deuxième acide aminé de la séquence).La séquence complète du tétrapeptide est: Cys-Glu-Arg-Tyr.
Soit
un hexapeptide obtenu par hydrolyse d'une protéine
1/ Comment peut on mettre en évidence la nature protéique du
composé obtenu ?
2/ Quelle procédure à suivre pour déterminer la formule
brute de l'hexapeptide ? Le résultat est: Ala1, Arg1, Leu1, Phe1,
Tyr1, Val1
3/ Déterminer la séquence du peptide, sachant que:
- L'aminopeptidase libère l'alanine
- L'hydrolyse par la trypsine conduit à deux tripeptides dont l'un
est constitué d'alanine, d'arginine et de tyrosine.
- L'hydrolyse acide partielle
conduit à un mélange de peptides: pepride P1, constitué
d'arginine et de phénylalanine; peptide P2 constitué de
leucine et de valine; peptide P3 constitué de phénylalanine,
leucine et arginine. En déduire la structure de l'hexapeptide.
1/
Nature protéique du composé (hexapeptide): On fait une réaction du biuret
2/Procédure à suivre pour déterminer la formule brute de l'hexapeptide: Hydrolyse acide totale puis électrophorèse et /ou chromatographie
d'échange d'ions.
3/Structure de l'hexapeptide
:
Ala-Tyr-Arg-Phe-Leu-Val
Etude du fragment N-terminal d'une protéine
Après
hydrolyse par la trypsine d'une protéine, le fragment N-terminal a été isolé. L'hydrolyse total de ce dernier donne Lys1, Arg1, Pro1, Asp1, Thr2, Glu2, Ser1, Gly1, Ala2, Val4, Leu1, Tyr3.
La réaction de Sanger donne un DNP-Lys.
L'hydrolyse par la chymotrypsine donne quatre peptides principaux dont deux, C1 et C2, sont basiques et ont pour composition:
- C1: Lys1, Pro1, Thr1, Val1, Leu1, Tyr1.
- C2: Arg1, Thr1, Ser1, Val1, Glu1.
Quelle est la position de C1 et C2 au sein du fragment N-terminal de la protéine
? Justifie.
L'attaque de C1 par la carboxypeptidase donne un acide aminé aromatique,
puis la leucine, puis un acide aminé neutre à cinq carbones.
L'hydrolyse partielle de C2 donne entre autres trois peptides dont les compositions
sont les suivantes: (Thr1, Glu1, Val1); (Ser1, Thr1, Val1); (Arg1, Ser1,
Val1).
Quelles sont les séquences de C1 et C2 ?
Position de C1 et C2 au sein du fragment N-terminal de la protéine:
C1 au début, C2 à la fin.
Séquences de C1 et C2: C1 = Lys-Pro-Thr-Val-Leu-Tyr, C2 = Glu-Thr-Val-Ser-Arg
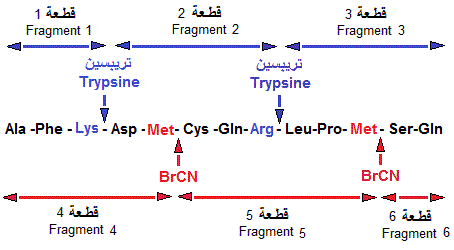
Soit un nanopeptide sujet à plusieurs analyses visant la détermination
de sa séquence en acides aminés. A la lumière des
résultats suivants, déduire la séquence du polypeptide.
- Hydrolyse acide: (Ala2 , Arg , Lys2 , Met, Phe, Ser2);
- Digestion à la carboxypeptidase A: Ala;
- Digestion à la trypsine: (Ala, Arg), (Lys, Phe, Ser), (Lys), (Ala, Met, Ser)
- Traitement au bromure de cyanogène: (Ala, Arg, Lys2 , Met, Phe, Ser), (Ala, Ser)
- Digestion à la thermolysine: (Ala), (Ala, Arg, Ser), (Lys2 , Met, Phe, Ser)
La digestion à la carboxypeptidase A nous indique que le résidu C-terminal du peptide est Ala.
La digestion à la trypsine nous permet d'ordonner partiellement deux des quatre fragments obtenus (rappel: la trypsine génère des fragments dont le C-terminal est Arg ou Lys):
Ala-Arg (Phe, Ser)-Lys
De plus, la digestion à la trypsine indique qu'une Lys est situé du côté C-terminal de Lys ou Arg (libération d'une lysine avec la trypsine).
La digestion à la trypsine indique aussi que le tripeptide (Ala, Met, Ser) correspond au Cterminal du peptide (il n'y a pas de Arg ou Lys dans ce fragment). De plus, la digestion au CNBr nous permet de déduire la position de la méthionine dans ce tripeptide comme étant:
Met-(Ala, Ser)
L'alanine étant le résidu C-terminal du peptide (voir digestion à la carboxypeptidase A), la séquence des trois résidus du C-terminal du peptide sera: Met-Ser-Ala
La thermolysine coupe du côté N-terminal de résidus hydrophobes: on peut donc déduire la position des résidus hydrophobes des deux fragments obtenus:
Ala-(Arg, Ser) Phe-(Lys, Lys,)-Met-Ser
Avec cette dernière information et considérant le patron
de digestion à la trypsine, on peut conclure que la séquence du peptide est:
Ala-Arg-Ser-Phe-Lys-Lys-Met-Ser-Ala
 |
 |
 |
La créatine
kinase préparée à partir du muscle possède une masse molaire d'environ 80000 g/mole. Elle est formée de deux sous-unités identiques. Elles est comparée à une protéine P de masse molaire 60000 g/mole de pHi supérieur et ne comportant qu'une seule sous unité.
Comparer le comportement des deux protéines dans les systèmes suivants:
1/ Eléctrophorèse sur papier.
2/ Chromatographi d'exclusion .
3/ Electrophorèse sur gel de polyacrylamide en milieu contenant du
dodécyl-sulfate de sodium (SDS) qui détruit la structure
quaternaire.
1/
Eléctrophorèse
sur papier:
La protéine P, possédant un pHi supérieur, sera chargé positivement dans un tampon de pH inférieur et migre vers la cathode (-). La créatine kinase migrera du côté anode (+),
car chargée négativement (si le pH du tampon d'électrophorèse est supérieur à son pHi)
2/ Chromatographie d'exclusion:
la séparation se fait en fonction de la taille des molécules. Les protéines à poids moléculaire élevé
sont collectées les premières. La créatine kinase,
dimère, sera éluée la première.
3/ Electrophorèse sur gel de polyacrylamide en présence de
SDS: comme le SDS dissocie les protéines en différentes
sous unités et les charge négativement, on sera en présence
de molécules de tailles 40000 (appartenant à la créatine
kinase) et une molécule de poids moléculaire 60000, chragées
toutes négativement. La séparation se fait en fonction de
la taille des molécules, uniquement. La créatine kinase
(2 monomères) migrent rapidement (plus loin) vers l'anode.
Structure de l'hémoglobine
L'hémoglobine
de l'Homme est constituée de 4 chaînes polypeptidiquesdont
deux chaînes alpha et deux chaînes béta (hémoglobine
normale HbA). Une fois l'hémoglobine
est mise en solution à pH 4,0, les chaînes se dissocient
comme suivant: alpha2-béta2 ------- Alpha2 + Béta2. Cette
dissociation est réversible. En effet, dès le rétablissement
du pH à la neutralité, l'hémoglobine reprend sa structure
alpha2-béta2. L'hémoglobine anormale HbC
diffère de HbA seulement par la présence sur les chaînes
alpha d'un résidu lysil à la place du résidu
glutamyl. HbI, une autre hémoglobine
anormale diffère de HbA par la substitution d'un acide aminé
par un autre. Lorsqu'on amène une solution de HbC et HbI à
pH 4,0 et on revient à la neutralité, on se rend compte
que la solution contient 4 types d'hémoglobine dont HbC, HbI, HbA
et un type moléculaire nouveau.
- Quelle est la chaîne mutée dans HbI ?
- Quelle est la formule du nouveau type d'hémoglobine ?
-
La chaîne mutée dans HbI est chaîne béta. En effet, HbC est alpha2C-béta2 et HbI est alpha2-béta2I. Ainsi, la dissociation complète à pH 4,0 permet de libérer alpha2C, alpha2, béta2 et béta2I. En revenant à la neutralité, toutes les recombinaisons entre alpha2 et béta2
sont réalisables:
alpha2C-béta2 (HbC), alpha2C-béta2I (type moléculaire nouveau), alpha2-béta2 (HbA) et alpha2-béta2I (HbI)
- Formule du nouveau type d'hémoglobine: alpha2C-béta2I
VIDEOS
Aspartame, vidéo narration en Français, sous-titrage en Arabe
LIENS UTILES
- Biochimie structurale (Contrôle final QCM corrigé, S3 (archives 2024)
- Protéines. Séquences (Exercices)
- Ionisation des acides aminés
- calcul du pHi d'un peptide
Afin de pouvoir continuer à servir les visiteurs, soutenez nos actions sur le site takween en faisant acquisition des ouvrages et supports pédagogiques desitinés à améliorer l'enseignement et la recherche scientfique en Biochimie.

Takween. Supports
Ouvrages (livres)
multilingues pour faciliter la transition Secondaire-Supérieur
- Livre
'Sciences de la vie. Protéines et Enzymes' (+ DVD), Baaziz, 2013:
QCM corrigés, Exercices corrigés, Contrôles corrigés
en Biologie cellulaire et Biochimie pour S1, S3, S4 et S5.

- Livre 'Sucres.Structures et
Métabolisme' Ar, Fr, Baaziz, 2018 (+ DVD), QCMs, Exercices: Ouvrage
facilitant la transition Secondaire-Supérieur (Ar, Fr)
Chaine
Youtube (abonnement). Plusieurs vidéos multilingues
 Enseignements par semestres
Enseignements par semestres
Congrès, Conférences,...
- Congrès, Conférences
- Cours, Workshops
- Bourses, Postdocs
- Biotech-ecolo annonces
الولوج إلى
الجامعة
ACCES A L'UNIVERSITE