

L'hémoglobine (خضاب، هيموغلوبين) est une structure 'alpha 2-béta 2'. Les sous unités sont constituées de chaînes polypeptidiques. Le site actif, comme montré par Rastop, comprend L'hème et deux histidines
4 pages: page 1, page 2, page 3 et page 4), les étudiants en Biochimie vont s'initier à la mise en évidence des structures protéiques en s'aidant du logiciel de visualisation 3D RasTop (structure Rastop).
| French | English | Arabic |
| Hémoglobine | Hemoglobin | خضاب، هيموغلوبين |
| Hème | Heme | يحمور، هيم |
- Hémoglobine. Page 2
- Hémoglobine. Page 3
- Hémoglobine. Page 4
- Hémoglobine. Préambule
---- VIDEO DE L'HEMOGLOBINE (Fr)
5/ Pour se focaliser sur des interactions non-covalentes spécifiques, comme les liaisons hydrogènes et électrostatiques ainsi que les interactions hydrophobes, on doit mettre en évidence certains résidus des acides aminés de la protéines.
Pour faciliter ce travail, on isolera une seule sous-unité de la protéine. Dans cet exemple, on étudie une sous-unité béta. Les 4 chaînes polypeptidiques sont notées
A, B, C et D. Pour avoir des informations sur la séquence des
acides aminés des 4 chaînes, saisissez 'show sequence'
dans la boite des commandes de RasMol, puis valider par la touche entrée.
La chaîne B est une sous-unité béta.
Pour travailler uniquement sur cette chaine, saisissez 'restrict
:B' dans la boite des commandes de RasMol, puis valider par la touche
entrée. saisir 'select hem' à cette étape, permet de visualiser les 4 hems.
Pour visualiser uniquement L'hème de la sous-unité B, saisir select hemB'. On obtient une image identique à la figure 1
6.
Pour sélectionner certains résidus d'acides aminés (ici, Histidine 92 = résidu His proximal de la chaine), on va saisir his92B dans le menu 'select atom epression (8e.bouton de la troisième rangée). On peut aussi saisir dans la boîte des commandes RasTop 'select his92B' puis 'color black' pour lui affecter une couleur noire. Sélectionner ensuite la visualisation sous 'Ball & Stick'. Réutiliser les commandes pour visualiser le résidu his63 (résidu His distal de la chaîne) et lui affecter
la couleur verte (green) dans un format 'Ball & Stick'.
Remarque: Saisir "select his92" selectionnera les 2 chaînes béta. Saisir 'select his' selectionnera toutes les His des 4 chaînes.
Pour éliminer les His des autres chaînes, saisir 'restrict:b' dans la boîte des commandes. On obtient une image correspondant à la figure 2

figure 2
7/ Pour montrer la relation entre les résidu His et L'hème, il faut visualiser l'atome de fer portée par L'hème. Pour cela, effectuer une rotation de l'image de façon à ce que l'atome de fer soit plus visible parmi les autres molécules. Pointer avec la souris sur l'atome de fer permet d'inscrire dans la boîte des commande RasTop, l'identification "Atom: Fe 4418 Hetero: Hem 147 Chain: B". Le fer est donc identifié comme l'atome N° 4418. Sélectionner l'atome en activant le bouton de selection des atomes (16e. bouton de la deuxième rangéee de la fenêtre). activer la palette des couleurs sur 'atoms' et selectionner la couleur de votre choix (ici bleu). Désactiver ensuite le bouton de sélection en faisant un nouveau clic dessus. Ausi, Le même résultat peut être obtenu en saisissant 'select atomno=4418' dans la boite des commandes de RasTop et lui affecter une couleur bleue (blue) (color blue). L'image obtenue correspond à la figure 3.

figure 3
8/ Pour marquer le nom d'un acide aminé, il faut activer le bouton de marquage (labels) (24e. bouton de la deuxième rangée de la fenêtre) et clique par la souris sur le motif à nommer. Le marquage peut être enlever en effectuant un deuxième clic sur le motif. Après, n'oublier pas de désactiver le bouton de marquage en faisant un nouveau clic dessus. Le même résultat peut être obtenu en saisissantla commande 'set picking label' dans la boîte des commandes RasMol, puis appuyer sur un atome du résidu qu'on désire marquer. En appuyant encore une fois sur le même atom, la marque sera enlevée. Pour désactiver la commande de marquage, saisir 'set picking ident'. L'image obtenue correspond à la figure 4.
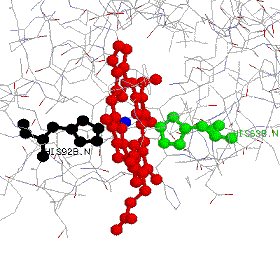
figure 4.




Hémoglobine. Structure 3D du complexe protéine-ligand (suite)
الهيموغلوبين. ثلاثي الأبعاد للمعقد بروتين ليجند
Vidéo sur l'Hémoglobine (Fr)
LIENS UTILES
-
Préambule
- Tous les Projets
- Projet 'Hémoglobine'
(Tutorial)
- Projet 'Lysozyme'
- Projet 'Transaminases'
- Projet 'Peroxydases'
- Hémoglobine.
Structure par JMOL

360 pages, 2018, ISBN : 978-9920-35-345-8 + DVD mis à jour + Assistance.

306 pages, 2013, ISBN : 978-99-54-32-663-3 + DVD mis à jour 2018 + Assistance
Comment avoir ces livres ? - أين يمكن اقتناء الكتب؟
Chaine Youtube (abonnement). Vidéos multilingues

-
من علوم الحياة إلى البيوكيمياء
Des Sciences de la vie au Lycée à la Biochimie à l'Université
-
تحميل ملفات في مواضيع البيوكيمياء
Téléchargement de fichiers en Biochimie
Afin de pouvoir continuer à servir les visiteurs, soutenez nos actions sur le site takween en faisant acquisition des ouvrages et supports pédagogiques desitinés à améliorer l'enseignement et la recherche scientfique en Biochimie.