L'extraction de l'ADN du plasmide est réalisée par lyse alcaline. Les plasmides sont utiles pour le clonage. Le séquençage de l'ADN peut être réalisé par la méthode de Sanger.
| French
------------ |
English
------------ |
Arabic
------------- |
| Plasmide | Plasmid | بلاسميد |
| Extraction de l'ADN | DNA extraction | إستخلاص ADN |
| Clonage | Cloning | إستنساخ |
Exercice 1 (Réponse 1). Interprétation de l'extraction de l'ADN plasmidique par lyse alcaline:
Les techniques les plus courantes de l'extraction et la purification
de l'ADN de plasmide par lyse alcaline ont des noms abrégés selon 'miniprep', 'midiprep' et 'maxiprep' (selon le volume de la culture bactérienne). Le but consiste à faire une extraction rapide de l'ADN du plasmide (ADN plasmidique) afin de faire une analyse par digestion via les enzymes de
restriction et la séparation par électrophorèse. Les digestions servent à vérifier un clone et/ou établir une carte de restriction.
Expliquer les différentes étapes de l'extraction de l'ADN plasmidique par lyse alcaline comme résumée dans la figure ci contre.

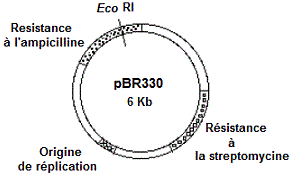
Exercice 2 (d'après https://step.ipgp.jussieu.fr) (Réponse 2). Clonage et analyse de l'ADN recombinant. On souhaite étudier la fonctionnalité d'un gène M d'une bactérie. Pour cela, on essaie de cloner au site EcoRI du vecteur plasmidique pBR330 (voir schéma) un fragment Eco RI-Eco RI d'ADN génomique de la bactérie d'intérêt.
a-
Proposez un protocole de clonage et indiquez comment vous sélectionnez les clones recombinants.
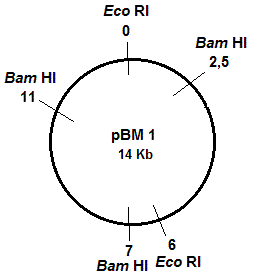
b- Un des plasmides recombinants contenant le gène M (appelé pBM1) est digéré par les enzymes de restriction Bam HI et Eco RI. Après migration et séparation des fragments d'ADN par électrophorèses sur gel d'agarose puis coloration au bromure d'éthidium, on obtient les profils de restriction ci contre.
Donnez la carte de restriction du plasmide recombinant pBM1.

Exercice 3 (Réponse 3). Carte de restriction d'un plasmide circulaire double brin.
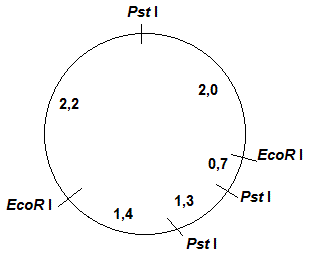
On se propose d'établir la carte de restriction d'un plasmide par deux enzymes de restriction, EcoR I et Pst I. La digestion par les deux enzymes a donné les fragments indiqués dans le tableau ci dessous. Dessiner la carte de restriction du plasmide.

 |
 |
 |
 |
Exercice 4 (Réponse 4). Séquençage de l'ADN d'un gène
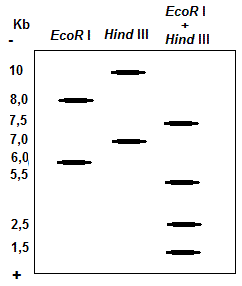
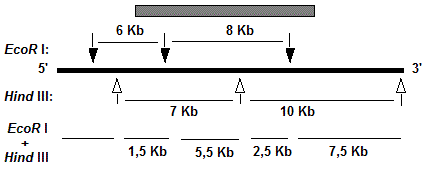
Pour établir la carte de restriction de l'ADN d'un gène, une solution d'ADN a été réparti en 3 fractions soumises à une hydrolyse avec EcoR I, Hind III et EcoR I + Hind III, respectivement. Les hydrolysats ont été séparés par électrophorèse.
La détection des fraguements d'ADN a été réalisée par hybrIdation avec une sonde marquée
du gène étudié. Les résultats de l'électrophorèse sont montrés dans la figure ci dessous. Dessiner la carte de restriction de l'ADN du gène.
Réponses
Réponse 1
(Exercice
1)
- Lyse des cellules par le détergent (SDS) en présence de
soude (pH 13) dénaturant l'ADN total (plasmide + Chromosome).
- Neutralisation rapide par addition de l'acétate de potassium (pH
5,5): L'ADN plasmidique se renature tandis que l'ADN chromosomique précipite.
- Concentration de l'ADN plasmidique par précipitation à l'alcool
et récupération de l'ADN par centrifugation.
- Suspension de l'ADN plasmidique dans le tampon TE ou l'eau pure.
- Elimination des ARN par les RNases (hydrolyse sélective des ARN/
ADN reste intact).
Réponse
2 (Exercice 2)
a. Protocole de clonage
- Préparation de l'insert: Extraction d'ADN génomique de
la bactérie. Digestion partielle de cet ADN par l'enzyme de restriction
Eco RI, de manière à générer des fragments
de 5 à 10 kb. Electrophorèse préparative pour purifier
sur gel les fragments de 5 à 10 kb.
- Préparation du vecteur: Digestion complète de pBR330 par
Eco RI. Déphosphorylation des extrémités.
- Clonage: ligature des inserts et des vecteurs. Transformation de cellules
d'E. coli. Recommencer les étapes précédentes
jusqu'à avoir 10 e+6 colonies transformées indépendantes.
- Sélection: sélection des colonies transformées
sur milieu contenant de la streptomycine. Pour estimer le taux de colonies
qui ont reçu un plasmide contenant de l'ADN génomique de
la bactérie étudiée, repiquage d'environ 500 colonies
sur milieu contenant de l'ampicilline: seule les colonies qui ont reçu
un plasmide ne contenant pas d'ADN génomique de la bactérie
étudiée poussent.
- Sélection des clones contenant le gène d'intérêt:
transfert des 10 e+6 colonies sur filtre et hybridation moléculaire
avec une sonde appropriée (technique dite de 'hybridation sur colonies')
b. Carte de restriction.
Une carte de restriction (= physique, à ne pas confondre avec
une carte génétique) est une représentatio
ngraphique de la localisation des sites reconnus par les principales
enzymes de restriction sur une molécule d'ADN. A l'aide
de la carte, il devient facile de connaitre la taille des fragments
d'ADN libérés par digestion par une ou plusieurs enzymes.
Rappel:
Un polylinker est une courte séquence portant un grand
nombre de site de restriction uniques sur l'ensemble du plasmide
et qui permettent d'insérer plus facilement un ADN d'intérêt
qu'on appelle insert. Le polylinker sert pour les manipulations
de génie génétique. Présentant un polylinker,
Un plasmide ou un ADN de bactériophage devient un vecteur
de clonage dans lequel on peut insérer un ADN étranger.
Exemple de Plasmide artificiel de clonage pBluescript

On note trois grands domaines fonctionnels: l'origine qui permet la réplication du plasmide, le gène 'ampicillin' qui code pour la résistance à l'antibiotique ampicilline et le gène lacZ qui code pour la ß-galactosidase et permet la détection des cellules transformées. Le site MCS (Multiple Cloning Site) ou polylinker, de courte séquence mais portant un grand nombre de sites de restriction uniques sur l'ensemble du plasmide, permet d'insérer facilement un ADN d'intérêt qu'on appelle insert.
Réponse 3 (Exercice 3). Carte de restriction d'un plasmide circulaire double brin.
EcoR I (2 coupures): 4,2 + 3,4 = 7,6
Pst I (3 coupures): 3,6 + 2,7 + 1,3 = 7,6
EcoR I + Pst I (5 coupures): 2,2 + 2,0 + 1,4 + 1,3 + 0,7 = 7,6
Réponse 4 (Exercice 4). Séquençage de l'ADN d'un gène..
Réponse 5 (Exercice 5). Séquençage d'un ADN monocaténaire
- Dans le séquençage par la méthode de Sanger, l'amorce
(primer) fourni une extrémité 3' OH libre nécessaire
pour l'action de la DNA polymérase.
- Le didésoxynucléotide provoque l'arrêt de l'élongation
de l'ADN.
- Le but du traitement thermique est la dénaturation de l'ADN bicaténaire
(Matrice + ADN néoformé). Après chauffage, tous les
fragments sont monocaténaires.
- Toutes les chaînes nucléotidiques liées à l'amorce
sont radioactives (32P) et visualisées par autoradiographie.
- La séquence de l'ADN à séquencer: 5'TCGAAGATGCACCTG3'
Automatisation du séquençage
Les fragments d'ADN résultant de la polymérisation sont marqués
grâce à des marqueurs fluorescents. La taille des fragments
obtenus est déterminée par une chromatographie au lieu
de l'électrophorèse. Le séquenceur détecte
la florescence sortant des colonnes de chromatographie (4 colonnes), repérant
les fragments d'ADN et leurs tailles précises. Les systèmes
les plus modernes permettent même de lire les quatres nucléotides
à partir d'une seule colonne de chromatographie.
Le résultat est présenté par la machine sous forme de courbes présentant la fluorescence détectée (et l'équivalent en terme de nucléotides).

Le séquençage des gènes par la méthode de Sanger permet d'étudier la phylogénie moléculaire des espèces.
Afin de pouvoir continuer à servir les visiteurs, soutenez nos actions sur le site takween en faisant acquisition des ouvrages et supports pédagogiques desitinés à améliorer l'enseignement et la recherche scientfique en Biochimie.

Takween. Supports
-Glossaire trilingues Français-Arabe-Anglais



Chaine
Youtube (abonnement). Plusieurs vidéos multilingues
 Enseignements par semestres
Enseignements par semestres
Congrès, Conférences,...
- Congrès, Conférences
- Cours, Workshops
- Bourses, Postdocs
- Biotech-ecolo annonces