|
French
|
Arabic
|
English
|
| Acides nucléiques | الأحماض النووية | Nucleic acids |
Les acides nucléiques englobent l'ADN et l'ARN
Exercice 1 (Réponse 1).
Pour une cellule de Mammifères, le poids moléculaire du DNA nucléaire natif est de 9.1011 daltons.
a/ Quelle est la longueur en pb et en mètre de ce DNA associé en double hélice (forme B)?
b/ La composition en bases de ce DNA montre un rapport A/G=4. Donner le pourcentage de chaque base pour ce DNA.
c/ Connaissant la Tm du poly-d-AT (69,3°C) et du poly-d-CG (110°C), quelle sera La Tm de ce DNA, dans les mêmes conditions expérimentales?
Exercice 2 (Réponse 2).
Soient deux DNA, A et B de 6000 pb. L'électrophorèse des deux DNA sur gel d'agarose 0,8% suivie d'une révélation par le bromure d'éthidium, a permis d'obtenir l'éléctrophorégramme 1. Après digestion des DNA par l'enzyme de restriction BamH I, l'électrophorèse donne l'électrophorégramme 2. La figure ci-dessous montre les résultats des électrophorèses. Pour rappel, une enzyme de restriction (endonucléase) coupure spécifiquement le DNA double brin au niveau d'une séquence bien déterminée.
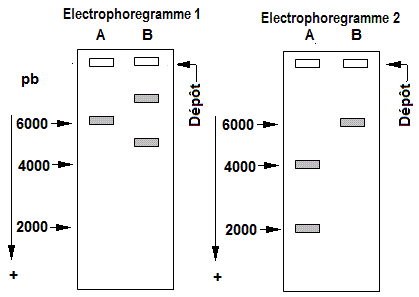
a- Commenter les profils électrophorétiques A et B des électrophorégrammes 1 et 2.
b- Quels seraient les profils électrophorétiques des deux DNA préalablement soumis à l'action d'une exonucléase avant l'électrophorèse.
c- Donner le profil électrophorétique qui serait obtenu si on fait agir une autre endonucléase de restriction pour laquelle chacune des molécules (A et B) possède deux sites de coupure distants de 4000 pb.
DNA A: AAGTTCTCTGAA
DNA B: GGACCTCTCAGG
DNA C: AGTCGTCAATGC
a- Donner les caractéristiques des DNA L, M et N relatives à la conformation (linéaire ou circulaire), simple ou double brin, PM, longueur en µm et nombre de nucléotides.
b- On chauffe les ADN L et M et on mesure leur absorbance à 260 nm. On obtient l'enregistrement ci-dessous. Expliquer la différence qui existe entre les deux profils à une température < 50°C. Interpréter les augmentations de la densité optique. Déterminer approximativement la Tm de L et M. Par quoi diffèrent les ADN L et M ?

c- Une solution N1 de l'ADN N a une absorbance de 2,8 à 25°C. En mélangeant 2,5 ml de N1 à 7,5 ml d'eau, on obtient la solution N2. Tracer sur le graphe précédent la courbe correspondant à N2.
Exercice 5 (Réponse
5).
On vise l'amplification génique in vitro d'un fragment d'ADN compris entre les nucléotides 5 et 100 (inclus) de la squence indiquée ci-dessous :

Trouver les séquences du couple d'amorces, chacune longue de 15 nucléotides.
On procéder à l'analyse d'un ARN. Sa composition globale en bases est 2A, 2C, 1U, 1G.
- Le traitement de l'ARN par la phosphodiestérase de venin de serpent libère pC.
- L'hydrolyse de l'ARN
par la ribonucléase pancréatique libère 1C, un
dinucléotide qui comprend A et C, et un trinucléotide
qui contient A, G et U.
- L'action de la ARNase T2 donne pAp, un dinucléotide qui contient
U et C, et un trinucléotide ayant A, G et C.
Quelle est la structure primaire de cet ARN?
On procède à l'analyse d'un ARN dont la composition brute en bases est 2A, 4C, 2G, 1U.
- La ribonucléase pancréatique libère 2Cp, deux dinucléotides dont un contient G et C et l'autre A et U, et un
trinucléotide fait de A, C et G.
- Un mélange de ARNase T1 et T2 produit C, Ap, pGp, et deux trinucléotides,
le premier contenant A et C et le deuxième CG et U.
- La phosphodiestérase du venin de serpent libère pC.
Quelle est la formule de cet ARN?
 |
 |  |
a-: Longueur de l'ADN = 1,5. 10 9 pb = 0,51 m;
b-: %C = %G = 10%et %A = %T = 40%.
c-: Tm = 77,44°C
a- L'ADN A peut correspondre à un ADN linéaire provenant d'un ADN circulaire (exemple ADN plasmidique) coupé sur les deux brins (suite à une longue conservation. L'ADN B peut correspondre à un ADN circulaire séparé selon ses conformations superenroulée (très mobile) et relachée (coupure sur un seul brin) avec une mobilité relativement faible. Voir électrophorèse de l'ADN des plasmides

Avec ces deux molécules séparées selon la forme, on ne peut connaitre le poids exact de l'ADN B. C'est la forme linéaire qui renseigne sur le poids réel de l'ADN.
L'électrophorégramme
B montre que les ADN A et B ont un seul site de restriction BamH I donnant deux fragments de 2000 pb et 4000 pb pour l'ADN A, car il est linéaire et un seul fragment de 6000 pb pour l'ADN circulaire B.
b- Si on considère l'action d'une exonucléase sur l' ADN A et l'ADN B, on n'aura
aucune bande pour l'ADN A, car il est linéaire. Il sera dégradé sur les extrémités. Par contre le profil de l'ADN B reste inchangé car il est circulaire et non accessible aux exonucléases.
c- ADN A linéaire: 3 fragments, car pour ADN linéaire, n coupures donnent n + 1 fragments
ADN B: circulaire: 2 fragments, car pour ADN circulaire, n coupures donnent n fragments
Tm ADN A < Tm ADN C < Tm ADN B
a: L est circulaire double brin, 388200 daltons, 0,22 micromètres.
M est linéaire,
double brin, 388200 daltons, 0,22 micromètres.
N est linéaire, simple brin, 970 nucléotides.
b: (M) = 2. (L),
effet hyperchrome, dénaturation thermique, Tm ADN L = 71°C,
Tm ADN M = 82°C,
%CG ADN L < %CG ADN M
c: DO 260 nm (N1) = 0,7
Séquence amorce
1: 5' AGATAAGTACTGTCA 3'
Séquence amorce 2: 5' ATGAGCATACATCTT 3'
La phosphodiestérase de venin de serpent ne coupe que le nucléotide situé en
3'-terminal de l'ARN, générant un nucléotide 5'-phosphate. Cette information nous indique que Cp est situé
en 3'-terminal de notre polynucléotide.
La ribonucléase pancréatique coupe l'ARN du côté
3' des nucléotides pyrimidiques, générant des produits de digestion se terminant par une pyrimidine 3'-phosphate. D'après
les résultats obtenus, on peut déduire les séquences
suivantes: 5'ACp3', 5'(A,G)Up3'
Finalement, la ribonucléase T2 coupe l'ARN après l'adénosine, générant des fragments se terminant avec une adénosine 3'phosphate. On peut donc utiliser cette information afin d'ordonner les séquences du produit obtenu: 5'(CG)Ap3'
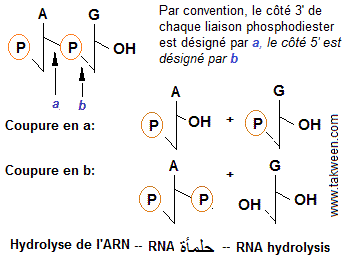
La présence de pAp après digestion avec la ribonucléase T2 indique que A est le nucléotide 5'-terminal.
En recoupant les informations obtenues par l'utilisation de ces trois enzymes, on obtient la séquence finale suivante: 5'pACGAUCp3'
Rappel: Les nucléases sont sélectives comme la plupart des enzymes. Elles sont spécifiques des molécules intervenant dans les réactions. Les ADNases (DNases) et les ARNases (RNases) sont spécifiques de l'ADN (DNA) et de l'ARN (RNA), respectivement.
La phosphodiestérase
de venin de serpent libère le nucléotide situé à l'extrémité 3' du fragment.
Le résultat obtenu nous indique que pC est le nucléotide 3' terminal pour notre polynucléotide.
La ribonucléase pancréatique coupe les molécules
d'ARN du côté 3' des pyrimidines, générant
des fragments se terminant avec une pyrimidine 3' phosphate. Grâce
aux fragments obtenus, on peut déduire en partie leur séquence:
5'GCp3', 5'AUp3', 5'(A,G)Cp3'
Le fait que l'on a libéré
2 Cp indique ces nucléotides sont situés tout de suite après U ou C.
L'ARNase T1 coupe du côté 3' de la guanosine, générant
des fragments se terminant avec une guanosine 3'phosphate. Quant à la ARNase T2, elle coupe du côté 3' des adénosine,
générant des adénosine 3'phosphate. L'utilisation combinée de ces deux enzymes donnera un mélange de fragments
coupés par l'un ou l'autre de ces enzymes, ou même par
les deux enzymes. On peut déduire la séquence des fragments
obtenus: 5'CCAp3', 5'(C,U)Gp3'
On a déduit la présence de deux C dans le premier fragment
car on mentionne qu'il s'agit d'un trinucléotide, et qu'il manque
un C si l'on fait le décompte des nucléotides obtenus
après digestion avec le mélange T1 + T2.
D'après le résultat obtenu avec la ribonucléase pancréatique, l'uridine est précédée de l'adénosine. On aura alors le fragment suivant: 5'AUCGp3'
De plus, toujours d'après le résultat avec T1 + T2, l'obtention de pGp indique que ce dernier
est le nucléotide 5'-terminal. Le résultat obtenu avec la ribonucléase pancréatique indique aussi que ce G est
suivi de C. Aussi, le fait d'avoir obtenu C et Ap indique que ces deux nucléotides sont situés tout de suite après A ou G.
On a donc jusqu'ici la séquence suivante: 5'pGC....AUCG....C3'
Il ne nous reste que A et C à placer. Comme avec la digestion
avec T1 + T2 on a eu la séquence 5'CCAp3', ceci suggère
que C est placé entre pGC...AUCG. Enfin, le dernier nucléotide,
A, devrait logiquement être situé à l'avant-dernière
place.
En tenant compte de ces différents résultats, on arrive à la séquence d'ARN suivante: 5'pGCCAUCGAC3'
| Enzyme | ADN, ARN, ADN + ARN | a ou b | Spécificité |
| Exonucléases | |||
| Phosphodiestérase du venin de serpent | ADN + ARN | a | Débute à l'extrémité 3', produit des XMP-5' |
| Phosphodiestérase du venin de la rate | ADN + ARN | b | Débute à l'extrémité 5', produit des XMP-3' |
| Endonucléases | |||
| RNase pancréatique | ARN | b | Libère des oligonucléotides à extrémité pyrimidine 3'-phosphate et des nucléosides 3'-phosphate pyrimidiques lorsqu'une pyrimidine est du côté 3' de la liaison diester. |
| RNase de Bacillus subtilis | ARN | b | Libère des oligonucléotides à extrémité purine 3'-phosphate et des nucléosides 3'-phosphate puriques lorsqu'une purine est du côté 3' de la liaison diester. |
| RNase T1 | ARN | b | Lorsqu'une guanine est du côté 3' de la liaison diester. |
| RNase T2 | ARN | b | Lorsqu'une adénine est du côté 3' de la liaison diester. |
| DNase I pancréatique | ADN | a | Coupe de préférence entre Py et Pu: incise l'ADN bicaténaire avec formation d'extremités 3'-OH |
| DNase II (Thymus, foie, Staphylococcus aureus) | ADN | b | Produit des oligpnucléotides |
| Nucléase S1 | ADN + ARN | a | Clive les acides nucléiques monocaténaires et non les acides nucléiques bicaténaires. |
Takween.
Supports
-Glossaire trilingues Français-Arabe-Anglais



Chaine
Youtube (abonnement). Plusieurs vidéos multilingues
 Enseignements par semestres
Enseignements par semestres
Cours, Workshops
Bourses, Postdocs
Biotech-ecolo annonces
CONCOURS
- Concours de Médecine

- Concours d'accès aux Etudes de Médecine dentaire
- Concours de pharmacie
-
من علوم الحياة إلى البيوكيمياء
Des Sciences de la vie au Lycée à la Biochimie à
l'Université
-
تحميل ملفات في مواضيع البيوكيمياء
Téléchargement de fichiers en Biochimie
Biochimie Calcul et Préparation des Concentrations. Vidéos explicatives